Heat maps (OMICS): tutorial en Excel
Este tutorial le mostrará cómo graficar e interpretar un mapa de calor (heat map) sobre datos OMICS en Excel usando el software estadÃstico XLSTAT.
Datos para ejecutar el análisis de mapa de calor en XLSTAT
Para este tutorial se utiliza una tabla de datos correspondiente a 1847 proteÃnas cuantificadas en 19 muestras extraÃdas de una hoja de maÃz y de acuerdo a 4 métodos de extracción basado en la proteómica âlabel-free shotgunâ (Langella et al. 2013). Estamos muy agradecidos a la plataforma PAPPSO (Gif-sur-Yvette, Francia), que nos proporcionó los datos y nos permitió usarlos para el tutorial. Las proteinas están almacenadas en filas y las muestras en columnas. Puede descargar aquà una hoja Excel con los datos y resultados.
Objetivo de este tutorial
El objetivo de este tutorial es utilizar la herramienta de análisis de datos exploratorios mapa de calor (heat map) para analizar agrupaciones de rasgos y de muestras simultáneamente de forma sintética. Asimismo, vamos a ser capaces de comprobar si los clusters de rasgos similares (proteÃnas en nuestro caso) corresponden a clusters de muestras similares. Si prefiere ver la versión de este tutorial en un video de 3 minutos, simplemente haga clic aquÃ.
Mapa de calor en XLSTAT: configuración del análisis
Para construir un mapa de calor en XLSTAT, haga clic en OMICs / Heat maps (Mapas de calor). En la pestaña General, seleccione la matriz de datos en el campo de la tabla Rasgos / Individuos. AquÃ, los individuos están representados por nuestras muestras. No necesita cambiar la opción Rasgos en filas, dado que las proteinas están almacenadas en las filas de la base de datos.
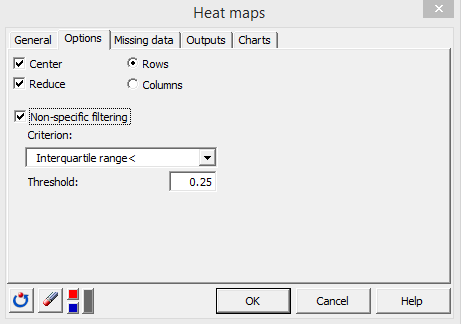
En la pestaña Opciones, activamos la opción Filtrado no especÃfico, seleccionamos Rango intercuartÃlico< y escribimos un umbral de, por ejemplo, 0.25. Esto eliminará todas las proteinas con un rango intercuartil inferior a 0.25 (i.e., con variabilidad baja). Asimismo contribuirá a mejorar la legibilidad del mapa de calor.
En la pestaña Datos perdidos, configuramos la estimación de datos perdidos usando el algoritmo Vecino más próximo.
En la pestaña Gráficos, elija escala de color del mapa de calor y juegue con las opciones anchura y altura para optimizar el tamaño del gráfico.
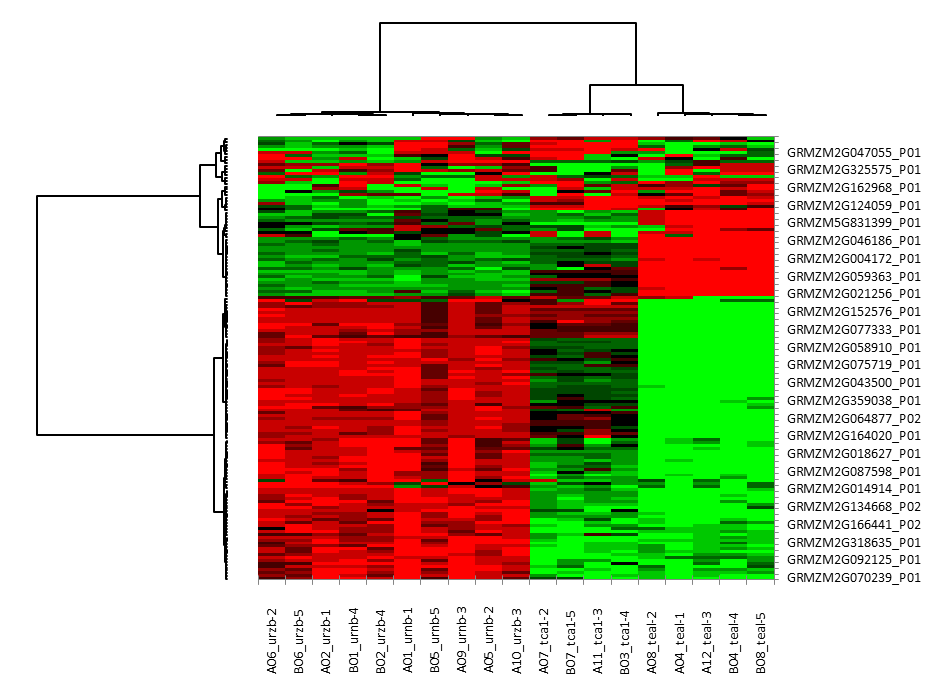
Mapa de calor (Heat map) en XLSTAT: análisis de los resultados
En primer lugar, vemos que el filtrado no especÃfico ha eliminado 1597 proteinas antes de llevar a cabo los cálculos para construir el mapa de calor.

Gráfico de mapa de calor: las proteÃnas se agrupan en filas y las muestras en columnas.
Si se analizan individualmente los dendogramas la muestra y la proteÃna, vemos claramente que:
-Â Â Â Â Â Â Las proteÃnas se dividen en dos grupos (dendrograma izquierdo) que corresponden aproximadamente a las proteÃnas especÃficas de la membrana y otras proteÃnas.
-      Las muestras se dividen en tres grupos (dendrograma superior). El cluster grande de la izquierda corresponde a las muestras cuantificadas con los métodos de extracción URZB y URNB. El cluster medio incluye muestras cuantificadas con el método TCA1 y el último corresponde al método TEAL.
El mapa representa los valores del conjunto de datos ordenados de nuevo de acuerdo con los dendrogramas.
Vamos a centrarnos en los patrones rectángulo / cuadrado dentro del mapa.
-      Los rectángulos grandes verdes y rojos de la izquierda muestran que para las muestras extraÃdas mediante los métodos URZB y URNB, tenemos una expresión relativamente alta del cluster superior de proteÃnas en comparación con el cluster inferior.
-      Por otra parte, las muestras de TEAL (parte derecha del mapa) muestran un patrón inverso de las cantidades de proteÃnas (relativamente baja para el cluster de proteÃnas superior y relativamente alta para el cluster inferior).
-      Finalmente, las muestras TCA1 (cluster medio) parecen exhibir cantidades intermedias en la mayorÃa de las proteÃnas (sin embargo, las proteÃnas de la parte inferior del mapa son relativamente más abundantes que las proteÃnas de la parte superior).
Referencia
Langella O, Valot B, Jacob D, Balliau T, Flores R, Hoogland C, Joets J, Zivy M(2013) Management and dissemination of MS proteomic data with PROTICdb: example of a quantitative comparison between methods of protein extraction, Proteomics. 2013 May;13(9):1457-66.
Â
Was this article useful?
- Yes
- No
 Help Center
Help Center