Expresión diferencial (OMICS): tutorial en Excel
Este tutorial le mostrará cómo configurar e interpretar un análisis de expresión diferencial en Excel usando el software estadÃstico XLSTAT.
Datos para ejecutar el análisis de expresión diferencial en XLSTAT
Para este tutorial hemos utilizado una tabla de datos simulados correspondientes a 36 muestras biológicas de individuos sanos y enfermos correspondientes a tres genotipos diferentes. Para cada muestra, se ha medido la expresión de 1561 gener a través de cuantificación RNA. Las RNA se almacenan en filas, y las muestras en columnas. A la derecha de la matriz de datos se han añadido un factor de genotipo y un factor de estado de salud. Los números de fila de los factores corresponden al número de muestras (número de columnas de la matriz de datos). Puede descargar aquà una hoja de Excel con los datos y los resultados.
Objetivo de este tutorial
El propósito de este tutorial es usar la herramienta de expresión diferencial de XLSTAT para identificar genes expresados diferencialmente de acuerdo con dos factores: genotipo (tres niveles: BB, BK y KK) y estado de salud (dos niveles: saludable y enfermo). Para cada factor, deseamos: 1)    Llevar a cabo un filtrado no especÃfico para eliminar los rasgos con variabilidad muy baja. 2)    Ejecutar automáticamente ANOVAs clásicos de una vÃa sobre cada una de los rasgos restantes y extraer los valores p. 3)    Corregir los valores p usando métodos apropiados para evitar efectos significativos por azar. Los rasgos (genes representados por las RNAs) asociadas a los valores p más bajos son aquellas que están más significativamente afectadas por el factor estudiado. Esta herramienta es muy útil para detectar conjuntos de genes que están vinculados a una enfermedad, por ejemplo. En el caso de factores de más de dos niveles (e.g., genotipo) podemos llevar a cabo múltiples comparaciones por pares para cada caracterÃstica. En el caso de factores de dos niveles (e.g., estado de salud), podemos generar gráficos de volcán para visualizar tanto las significaciones estadÃsticas como las biológicas asociadas a todos los rasgos. Advierta que la herramienta de expresión diferencial en XLSTAT puede asimismo usarse para estudiar los efectos de variables explicativas en la producción de proteinas o en la regulación metabólica en un contexto de datos OMICs de alto rendimiento.Â
Expresión diferencial en XLSTAT: configuración de los análisis
Para llevar a cabo el análisis de expresión diferencial, haga clic en XLSTAT-OMICs / Expresión diferencial. En la pestaña General, seleccione la matriz de datos en el campo de la tabla Rasgos / individuos. AquÃ, los individuos están representados por nuestras muestras. No es preciso modificar la opción Rasgos en filas, ya que los genes están almacenados en filas en la base de datos. Es obligatorio seleccionar la primera columna de la base de datos que contiene la caracterÃstica IDS. XLSTAT necesita esta información para que el usuario pueda identificar rasgos interesantes con sus nombres en la salida del análisis. En el campo de las variables explicativas, seleccione las dos columnas que contienen la afiliación de cada muestra a los niveles de los factores.
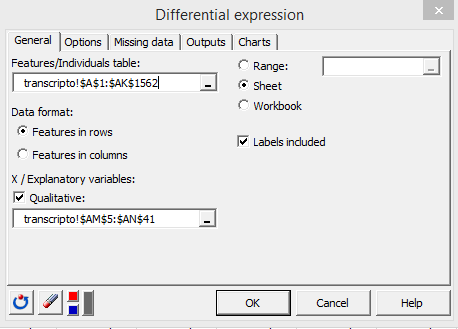
En la pestaña Opciones, seleccione el tipo de prueba Paramétrica. Esta opción producirá un ANOVA de una vÃa por factor y por caracterÃstica. Para números de muestra pequeños, recomendamos en cambio usar el método no paramétrico, que reemplaza los ANOVAs de una vÃa por pruebas de Kruskal-Wallis. En las correcciones post-hoc, elija el procedimiento Benjamini-Hochberg, que es comúnmente utilizado en estudios de expresión diferencial. Es parte de la familia de correciones en el valor p de la FDR (False Discovery Rate). Es muy adecuado en estudios que implican el cálculo de un número grande de valores p, dado que es menos estricto que las correcciones que son parte de la familia FWER (Family Wise Error Rate), tales como la corrección de Bonferroni. Ajuste el número de valores p para mantener a 30, para evitar que se muestre una lista enorme de valores p en los resultados (valores p altos no son en absoluto interesantes en el contexto de nuestro estudio). Active la opción comparaciones múltiples por pares y seleccione Tukey(HSD) para obtener comparaciones múltiples por pares entre los niveles de genotipo para cada gen. Finalmente, active la opción de filtrado no especÃfico, elija %(Desv. est.) con un umbral del 50% para eliminar al 50% de los genes basándose en el criterio de desviaciones estándar más bajas antes de los análisis.
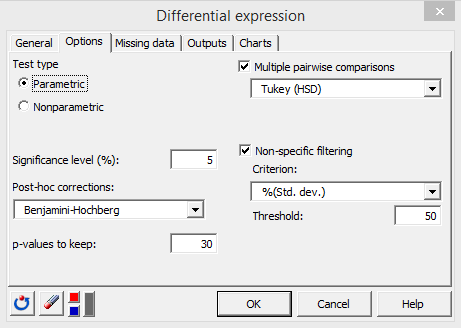
En la pestaña Gráficos, active las opciones Histograma de los valores p y Gráfico de volcán (Volcano plot).
Las dos opciones siguientes representan dos formas de representar los efectos biológicos en el eje x del gráfico de volcán. Elegiremos Log2(razón de las medias), porque nuestros datos no están transformados. Active la opción Identificar rasgos. XLSTAT usará por tanto un color especial para los rasgos altamente significativas tanto en la escala estadÃstica como en la biológica, y de acuerdo con los dos umbrales siguientes. Elija 1 para Umbral(x). Un log2(razón de medias) de 1 significa que la media del numerador es dos veces la media del denominador. De forma inversa, un log2(razón de medias) de -1 significa que la media del denominador es dos veces la media del numerador. Un log2(razón de medias) de 2 o de -2 representa un FOLD cambio de 22, y asà sucesivamente. Elija un umbral de valor p de 0.001 en la ventana Umbral(y). Esto significa que el umbral de significación estadÃstica será âlog10(0.001).
Haga clic en el botón OK.
Expresión diferencial en XLSTAT: interpretación de los resultados
Después de un resumen sobre las diferentes opciones usadas en el análisis, aparecerá el número de rasgos que fueron eliminadas por el filtrado no especÃfico. A continuación se muestra un análisis por factor.
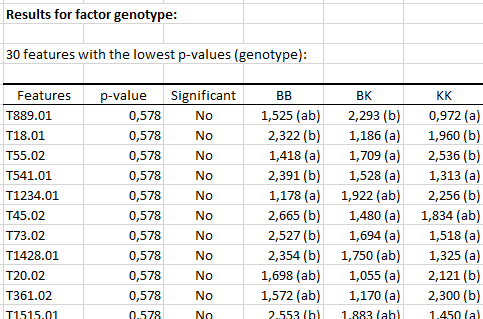
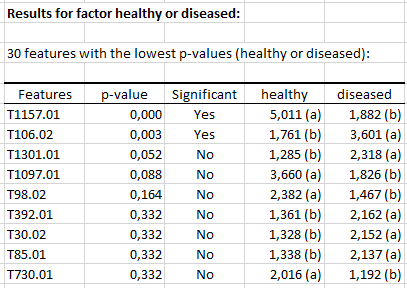
En primer lugar, se ofrece una tabla que muestra las 30 rasgos más significativos ordenados de acuerdo con los valores p en orden ascendente. La tabla contiene el nombre de la caracterÃstica o rasgo, los valores p penalizados, el nivel de significación, y las medias de cantidad RNA para cada nivel del factor. Si un valor p es significativo, el usuario puede estar interesado en llevar a cabo comparaciones múltiples por pares representadas por las letras asociadas a las medias. Dos niveles que comparten la misma letra no son significativamente diferentes. Dos niveles que no comparten la misma letra son significativamente diferentes.
En el caso del factor genotipo, no hay valores p significativos con alfa = 0.05. En este caso, interpretar comparaciones múltiples no es relevante para ninguno de los rasgos.
El histograma de los valores p muestra que están distribuidos de forma homogénea.
El factor de estado de salud (sano o enfermo) parece afectar a la expresión de dos genes: T1157.01 y T106.02. El primero tiene una mayor expresión en muestras sanas, y el segundo tiene una mayor expresión en las muestras enfermas.
Ambos rasgos pueden visualizarse en el gráfico de volcán (volcano plot):
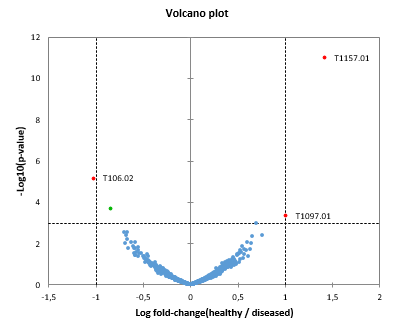
Los rasgos situados en las esquinas superior izquierda y superior derecha de la gráfica están etiquetados. Se corresponden con los rasgos que se apoderan de los umbrales de significación biológica y estadÃstica (lÃneas discontinuas).
Observe que los valores p usados para calcular âlog10(p-values) en el gráfico de volcán son los valores p brutos, no corregidos.
Was this article useful?
- Yes
- No
 Help Center
Help Center